Introducción
Las introducciones de especies son un fenómeno en constante
crecimiento a nivel global, con una tendencia exponencial que se acentuó a
mediados del siglo XX (Seebens et al. 2017). Sin
embargo, no son un fenómeno nuevo, ya que los humanos hemos transportado
especies a través de barreras biogeográficas durante milenios, tanto domésticas
como silvestres (Wilmshurst et al. 2008; Hofman y Rick 2018; Peters et
al. 2022). Como resultado, un número desconocido de organismos pudo haber
sido introducido en diversas áreas del mundo antes de que existieran
descripciones de las biotas y demasiado temprano como para haber dejado una
huella cultural que permita reconocer la introducción en la actualidad (Haydar 2012). Estos taxones pueden estar socialmente
aceptados como organismos nativos, generando una forma particular del síndrome
de las referencias cambiantes (shifting baseline syndrome) (Clavero 2014), o su origen puede permanecer rodeado de
incertidumbre, siendo entonces denominados especies criptogénicas (Carlton 1996).
Que una especie sea nativa en un territorio, es decir, que
su presencia actual y/o histórica no haya requerido intervención humana activa,
es un dato fundamental para planificar la conservación de la biodiversidad (Oficialdegui et al. 2024). Sin embargo, a menudo
resulta complejo desentrañar el estatus de especies que pudieron haber sido
introducidas en tiempos remotos. La evidencia arqueológica puede proporcionar
información útil para datar la llegada de un taxón a un lugar (Detry et al. 2018), mientras que en ocasiones existen
documentos históricos que incluyen pruebas directas de eventos de introducción
y sus motivaciones (Pipek et al. 2015; Clavero 2022). Los análisis de patrones biogeográficos y
la presencia de organismos asociados (comensales, parásitos) también pueden
proporcionar información clave para determinar el estatus de un taxón (Blakeslee et al. 2008; 2012).
En las últimas décadas, se ha generalizado el uso de análisis moleculares,
principalmente basados en marcadores genéticos, para evaluar el origen de
poblaciones cuyo estatus (nativo o no) resulta dudoso.
Describir introducciones históricas a través de narrativas
sólidas requiere integrar resultados obtenidos desde diversas disciplinas (Szabó 2010; Craven et al. 2019).
Cuando surgen conclusiones contradictorias, es necesario revisar las
interpretaciones obtenidas desde los diferentes enfoques, evitando otorgar a
priori la preeminencia de unos sobre otros. Esto es particularmente
relevante en los resultados basados en ADN, ya que se tiende a asumir, tanto
por parte de la comunidad científica como por el público en general, que éstos
son más informativos que los obtenidos a través de aproximaciones más
tradicionales, como el estudio de fósiles o de documentos históricos (para
prejuicios sobre diferentes disciplinas, ver Pooley et
al. 2014). A menudo se pasa por alto que las conclusiones derivadas de los
análisis genéticos están mediadas por la interpretación de los resultados que,
como en cualquier otro enfoque científico, tiene asociadas limitaciones. En
este trabajo discutimos cómo los datos genéticos (principalmente marcadores de
ADN mitocondrial) deberían interpretarse, y cómo no debería hacerse, para
determinar si un taxón es nativo en un territorio. Para ello, revisamos y
describimos las limitaciones que la información genética puede tener en la
reconstrucción de movimientos de especies en el pasado.
Como ejemplo, desarrollamos el caso del cangrejo de río
italiano (Austropotamobius
fulcisianus, en adelante, cangrejo italiano), una especie
introducida en la península ibérica en el siglo XVI (Clavero
2022). Durante el siglo XX, se hizo una intensa promoción de la explotación
de esta especie y, tras su colapso en la década de los 1970s, se ha trabajado
para su conservación (Clavero 2016). Aunque la
introducción del cangrejo italiano y el subsecuente proceso de invasión se
habían descrito integrando evidencias obtenidas por diversas disciplinas (Clavero et al. 2016), distintos estudios basados en ADN
han venido afirmando que la especie es en realidad nativa de la península
ibérica (por ejemplo, Matallanas et al. 2016; Martínez-Ríos et al. 2023). En la parte final del
artículo, utilizamos resultados ya publicados para demostrar que la
variabilidad de las secuencias de ADN se ha interpretado erróneamente en
distintos trabajos y que, de hecho, los patrones genéticos refuerzan la idea de
que el cangrejo italiano es una especie introducida en la península ibérica.
Haplotipos: métricas y redes
Los trabajos que usan marcadores genéticos para discernir si
una especie es nativa a menudo se fijan en indicadores de diversidad genética
(por ejemplo, la riqueza de haplotipos) y exclusividad (es decir, la presencia
de haplotipos privados, no presentes en el área nativa) (Cunningham
2008). Sin embargo, ninguno de estos indicadores puede vincularse de forma
unívoca con el estatus nativo o no nativo de un taxón en un territorio (Uller y Leimu 2011).
La idea de que las poblaciones no nativas deberían tener una
baja diversidad genética se basa en la suposición de que las etapas de
transporte, introducción y establecimiento del proceso de invasión (sensu
Blackburn et al. 2011) conllevan un severo cuello
de botella genético, que dejaría una huella detectable en las poblaciones
introducidas. Esta suposición puede ser válida cuando se produce un único
evento de introducción (o muy pocos) que involucra pocos individuos (es decir,
cuando la presión de propágulo es baja). En esas condiciones tiene lugar un
efecto fundador, seguido de una deriva genética en la fase de expansión, que
resulta en una pérdida de diversidad genética (Ascunce
et al. 2011; Bélouard et al. 2019; Sequeira et al. 2022; Lambea-Camblor
et al. 2023) (Fig. 1A). Pero este es solo uno
de los muchos escenarios posibles en relación con las introducciones, y existen
numerosas opciones de generar poblaciones introducidas con una alta
variabilidad genética (Cabezas et al. 2014), como se
ejemplifica en la Figura 1.
Hace casi 20 años, Roman y Darling (2007) demostraron que la
diversidad genética de las poblaciones introducidas está determinada por i) el
acervo génico de las poblaciones fuente, ii) el número de eventos de
introducción, y iii) el tamaño del propágulo. Está bien descrito que la
diversidad genética en poblaciones introducidas puede ser alta, incluso
superando los valores observados en poblaciones nativas (Lejeusne
et al. 2014). Esta situación es especialmente frecuente cuando el propágulo
original proviene de diferentes poblaciones fuente (Blumenfeld
et al. 2021) (Fig. 1C) o de poblaciones
mezcladas (admixtured; i.e., generadas, dentro del área
nativa, a partir de individuos de diversos orígenes, Rius
y Darling 2014; van Boheemen et al. 2017) (Fig. 1D). Estas poblaciones que combinan
artificialmente características genéticas diversas son comunes en especies
utilizadas intensamente por el ser humano (por ejemplo, especies ornamentales,
de pesca recreativa o de acuicultura) (Barbaresi et
al. 2007; Oficialdegui et al. 2019).
Independientemente de la diversidad genética de poblaciones fuente, cuanto
mayor sea el número de eventos de introducción, mayor será la probabilidad de
encontrar más diversidad genética en las poblaciones resultantes (Dlugosch y Parker 2008; Facon
et al. 2008), especialmente si los eventos de introducción en un área
provienen de diversas poblaciones (Fig. 1C).
Incluso introducciones repetidas desde la misma fuente aliviarían las
reducciones en diversidad genética asociadas al efecto fundador y la deriva
genética (Fig. 1B) (Uller
y Leimu 2011). Las introducciones múltiples también son más frecuentes en
especies vinculadas a los humanos, como plantas ornamentales, especies de
acuicultura o mascotas, ya que los escapes y liberaciones (i.e.,
los eventos de introducción) a menudo ocurren en repetidas ocasiones (Kelager et al. 2013; Hunter y
Nico 2015; Larroque et al. 2023). Finalmente,
aun siendo iguales el resto de las características, cuanto mayor sea el número
de individuos involucrados en los eventos de introducción, mayor será la
probabilidad de encontrar una diversidad genética alta de las poblaciones
resultantes (Hufbauer et al. 2013). En este
sentido, el transporte de numerosos individuos es normalmente más frecuente en
introducciones intencionadas, dirigidas a establecer poblaciones viables, que
en introducciones involuntarias (Blackburn et al. 2013).
Dejando de lado la diversidad genética, el hecho de que en
poblaciones criptogénicas se detecten haplotipos privados se ha asociado a un
supuesto carácter nativo (Cunningham 2008). Este
razonamiento asume que si ciertos haplotipos se detectan únicamente en un área
es porque habrían surgido allí, lo que indicaría que la especie es nativa de
ese territorio. Consideramos que este argumento es erróneo porque la
exclusividad de los haplotipos depende en gran medida de la exhaustividad del
conocimiento sobre diversidad genética recabado en el área nativa (Muirhead et al. 2008; Fig. 1),
y a menudo es difícil garantizar que se haya realizado un muestreo completo de
los haplotipos existentes en ella. De hecho, dado que las especies invasoras
tienden a ser más estudiadas en lugares en los que no son nativas (Hierro et al. 2005), la probabilidad de detectar
haplotipos y considerarlos privados puede ser incluso mayor en éstos. Cabe
destacar que este sesgo puede favorecer la percepción de que la diversidad
haplotípica es mayor en ciertas áreas no nativas, lo que subraya la necesidad
de estandarizar la riqueza haplotípica en función del esfuerzo de muestreo,
tanto a nivel poblacional como en el número de individuos. Por ejemplo, Martínez-Laiz et al. (2021) describieron que más
de dos tercios de los haplotipos encontrados en poblaciones no nativas del
anfípodo Caprella
scaura eran privados, debido a que gran parte de la variabilidad
genética de la especie en su área nativa es desconocida.
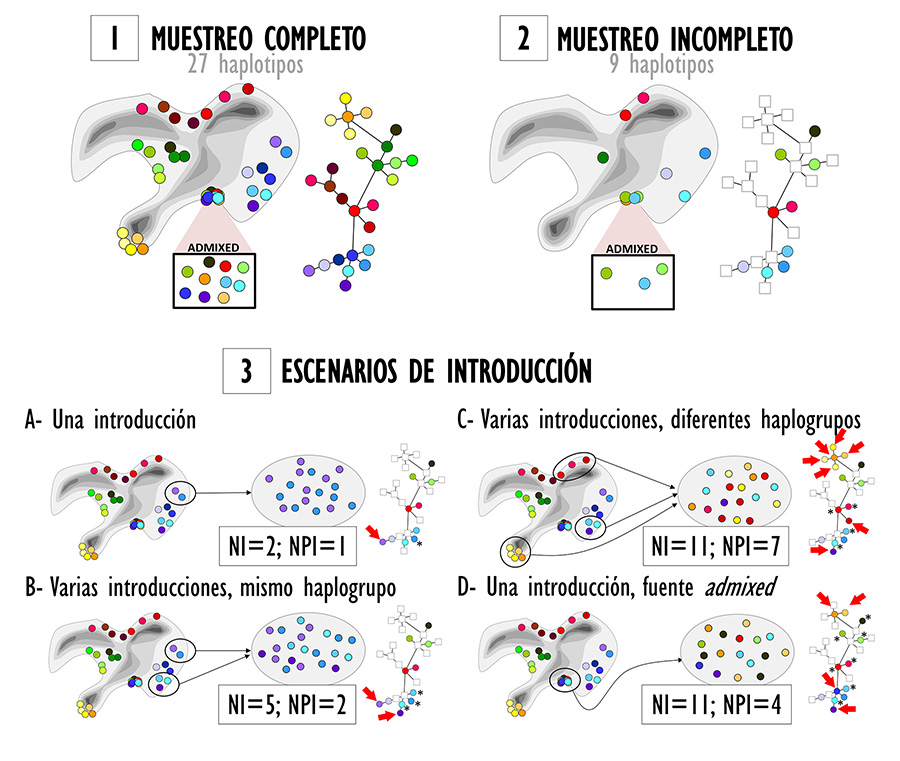
Figura 1. Posibles patrones de diversidad
genética de una hipotética especie en su área nativa y no nativa, bajo
diferentes escenarios de invasión, ejemplificando los mecanismos por los cuales
la diversidad genética puede ser alta en áreas no nativas. En 1 y 2 se representa
la situación en el área nativa. En 1, la variabilidad genética de las
poblaciones nativas ha sido estudiada en profundidad, identificando todos los
haplotipos (27 en total), cuyo sistema de relaciones se muestra, a la derecha,
en una red de haplotipos hipotética. El rectángulo inferior amplía un área
donde se encuentran poblaciones mezcladas (admixed). Sin embargo, esta
cobertura completa no es una situación común, por lo que en 2 se presenta un
caso en el que el conocimiento de la diversidad genética mucho más limitado (en
el ejemplo, solo se identifican 9 haplotipos y la red de haplotipos es notablemente
incompleta). En 3 se muestran posibles patrones de diversidad genética (NI =
número de haplotipos, NPI = número de haplotipos privados) y estructura (redes
de haplotipos) en áreas no nativas bajo diferentes escenarios de invasión,
variando el número de eventos de introducción y sus poblaciones de origen. En
las redes de haplotipos, los asteriscos (*) marcan los encontrados tanto en
áreas nativas como no nativas, y las flechas rojas indican haplotipos
exclusivos del área no nativa, asumiendo un muestreo incompleto del área nativa
(i.e., 2).
Figure 1.
Hypothetical genetic diversity patterns of a species within its native and
non-native ranges, under different possible invasion scenarios, exemplifying
mechanisms by which genetic diversity can be high in non-native areas. 1 and 2
represent the situation in the native area. In 1 native population and their
genetic pools have been thoroughly studied, so that all haplotypes (27 in
total) have been identified and their relationships is described by the
hypothetical haplotype network shown to the right. The lower rectangle
amplifies an area where admixed populations of the species are found. The full
coverage shown in 1 is not a common situation and knowledge of the genetic pool
is often incomplete due to sampling limitations, as shown in 2 (9 haplotypes
identified and notably incomplete haplotype network). 3 shows possible genetic
diversity (NI= number of haplotypes, NPI= number of private haplotypes) and
structure (networks) outcomes in non-native areas from different invasion
scenarios, varying the number of introduction events and their source
populations. In the networks, asterisks (*) mark haplotypes found in both
native and non-native areas and red arrows indicate those private to non-native
area, assuming an incomplete survey of the genetic pool in the native are (i.e.,
2).
En cualquier caso, es también posible que existan haplotipos
realmente privados en áreas no nativas. Por ejemplo, pueden haber surgido
nuevas variantes genéticas después de las introducciones, especialmente en
aquellas que hayan ocurrido mucho tiempo atrás (Einfeldt
et al. 2020). Además, como desarrollaremos más adelante, los haplotipos
encontrados en las áreas no nativas de un taxón pueden haber desaparecido del
área nativa original, generando una errónea percepción de exclusividad.
Las posibles interpretaciones erróneas de la riqueza o la
exclusividad haplotípica ocurren cuando los haplotipos se consideran unidades
independientes, en lugar de analizar las relaciones entre sus secuencias (es
decir, la estructura genética), sobre las que informan las redes o árboles de
haplotipos. Blakeslee et al. (2008) demostraron
que los vínculos entre haplotipos proporcionan una base sólida para evaluar si
una especie es nativa de un territorio. Si la especie ha colonizado un
territorio de forma natural y ha evolucionado allí, los haplotipos descritos en
él deberían formar un grupo coherente e independiente de los observados en
otras zonas nativas. Este es el patrón observado en la población ibérica de
rabilargo (Cyanopica),
un género de aves con una distribución vicariante, que incluye un núcleo
ibérico y otro en el oriente de Asia. Este peculiar patrón biogeográfico llevó
a proponer que el rabilargo hubiese sido introducido en la península ibérica,
pero esta posibilidad se descartó tras analizar el gen COI y descubrir dos
grupos de haplotipos separados (ibérico y asiático; Fok et
al. 2002). Sin embargo, si los haplotipos detectados en dos zonas distantes
y/o aisladas aparecen entremezclados en las redes o árboles cabe deducir que el
taxón ha sido introducido en una de ellas (Einfeldt et
al. 2014), incluso cuando algunos haplotipos solo se hayan detectado allí.
Blakeslee et al. (2008)
estudiaron Littorina
littorea, una especie de caracol propia de costas rocosas, cuyo
estatus en América del Norte era incierto, y encontraron que los haplotipos
mitocondriales descritos allí aparecían anidados dentro de haplotipos europeos
en las redes. A pesar de identificar un gran número de haplotipos en América
del Norte (n = 57) y que más del 50 % de ellos eran privados, los
autores concluyeron que la estructura genética apoyaba el estatus no nativo de L. littorea
en América del Norte, hipótesis que ya había sido sugerida anteriormente en
base a evidencias arqueológicas, históricas y biogeográficas (Blakeslee 2007). De manera similar, Trucchi y Sbordoni (2009) analizaron los
patrones genéticos del puercoespín (Hystrix cristata) en Italia y el
norte de África, mostrando que los 7 haplotipos detectados en Italia (de 161
especímenes) eran privados, pero aparecían entremezclados en las redes con los
6 haplotipos (de solo 6 individuos) identificados en el Magreb. Los autores
concluyeron que estos resultados respaldaban el estatus no nativo del
puercoespín en Italia y estimaron que habría habido diferentes eventos de
introducción, que, en base a diversas evidencias, Masseti
et al. (2010) sitúan en la alta edad media.
Problemas insalvables
Incluso aunque los datos genéticos hayan sido recopilados
minuciosamente y se analicen e interpreten correctamente, pueden ofrecer una
visión errónea sobre el estatus de una especie en un territorio. Esto se puede
deber a distintos factores, como la pérdida de la estructura genética en el
área nativa debido a introducciones realizadas dentro de ella (por ejemplo, Einfeldt et al. 2020), la incertidumbre o el
desconocimiento sobre el área de distribución nativa de una especie (como en la
hormiga Pheidole
megacephala, una especie invasora en muchos lugares del planeta; Fischer y Fisher 2013), o incluso porque especies
aún no descritas sean detectadas por primera vez como invasoras (Clavero et al. 2023). Sin embargo, el obstáculo más
común para evaluar el carácter nativo mediante marcadores genéticos es la
erosión genética en el área nativa causada por extinciones locales o regionales
(Marchetti y Engstrom 2016). No es raro que
una especie tenga un estado de conservación desfavorable en su área nativa
mientras prospera como invasora en otras regiones (Baquero
et al. 2023; Tedeschi et al. 2024), un fenómeno
que ha sido denominado la "Paradoja Conservación-Invasión" (Conservation-Invasion
Paradox, Hong et al. 2024). En casos
extremos, una especie puede estar extinta en su área nativa mientras sigue
existiendo —junto con parte de su acervo genético original, ahora único—
exclusivamente en áreas donde había sido introducida (Crain
y Moyle 2011).
El declive de una especie
a menudo lleva aparejada una pérdida de diversidad genética, que se hace
evidente cuando se comparan muestras contemporáneas e históricas (Díez-del-Molino et
al. 2018). Se han documentado
pérdidas importantes de haplogrupos o linajes ligados a declives severos en
varios taxones, como el íbice alpino (Capra ibex) (Robin et al. 2022) o el lince ibérico (Lynx
pardinus) (Casas-Marcé et al.
2017). Podría, por tanto, darse
el caso de que en el pasado ocurriesen introducciones que tuvieran como fuentes
poblaciones hoy desaparecidas, con haplotipos incluidos en linajes igualmente
inexistentes en el área nativa. En este escenario, los patrones genéticos
actuales podrían sugerir erróneamente que esas poblaciones introducidas son
únicas y han estado aisladas durante mucho tiempo, lo que llevaría a pensar que
son nativas de los territorios que hoy ocupan. Aunque pueda parecer
inverosímil, este tipo de situaciones han sido documentadas en diferentes
especies. Por ejemplo, el ciervo corso (Cervus elaphus subsp.
corsicanus) representa un linaje
único y muy divergente dentro del complejo específico del ciervo rojo. Se
encuentra exclusivamente en Córcega y Cerdeña, y se originó a partir de una
introducción antigua desde el centro de Italia (Hajji et al. 2008; Doan et al. 2017). De manera similar, Mallorca alberga una población introducida de
conejo (Oryctolagus cuniculus) que es genéticamente exclusiva y que divergió de otras poblaciones
ibéricas hace más de 100 mil años (Seixas et al. 2014).
Si nos fijásemos exclusivamente en los patrones genéticos
contemporáneos, tanto el ciervo corso como algunos de los conejos mallorquines
podrían ser considerados especies nativas en sus respectivas islas. Sin
embargo, contamos con un extenso conocimiento interdisciplinar (arqueológico,
geológico, histórico y biogeográfico) que demuestra que tanto los ciervos como
los conejos fueron introducidos en las islas mediterráneas. Estas islas se
separaron de las costas continentales hace aproximadamente 5 millones de años (Gippoliti y Amori 2006) y sabemos que los conejos
llegaron a Mallorca solo después de la colonización humana de la isla, hace
aproximadamente 4300 años (Alcover 2008). Cabe
resaltar que la información sobre los linajes exclusivos del ciervo y del
conejo en Córcega y Mallorca, respectivamente, no llevó a cuestionar el sólido
conocimiento acumulado sobre la evolución de las biotas insulares y el papel de
los humanos en su transformación. En cambio, los patrones genéticos observados
fueron contextualizados críticamente dentro de los procesos ecológicos y
antropogénicos ya conocidos. Sin embargo, este no es siempre el procedimiento
seguido en los estudios genéticos, que en ocasiones ignoran otras fuentes de
conocimiento.
El cangrejo italiano en España
Austropotamobius, uno de los tres géneros de
cangrejos de río nativos de Europa, tiene dos linajes principales, que
divergieron hace entre 10 y 15 millones de años (Klobučar
et al. 2013; Jelić et al. 2016). Estos linajes se
han considerado tradicionalmente dos especies, A. pallipes y A.
torrentium (Grandjean et al. 2002), pero
actualmente se consideran complejos específicos, cada uno de los cuales incluye
diferentes taxones con categoría de especie o subespecie (Pedraza-Lara et al. 2010; Parvulescu
2019). Dentro del complejo pallipes se aceptan actualmente dos
especies, el cangrejo de patas blancas (A. pallipes, sensu stricto)
y el cangrejo italiano (A. fulcisianus) (Crandall
y De Grave 2017). Ha existido cierta confusión respecto al nombre
científico del cangrejo italiano, ya que en base a los trabajos de Faxon (1914) se ha usado frecuentemente Austropotamobius
italicus (por ejemplo, Alonso 2011). Sin embargo,
el nombre A. fulcisianus, propuesto a finales del siglo XIX como Astacus pallipes var. fulcisiana (Ninni, 1886), debe
prevalecer por ser anterior (Crandall y de Grave 2017). En su descripción, Ninni usó el cangrejo
de río italiano (gambero fluviale italiano) como nombre común (Ninni 1886), empleo que mantenemos aquí (ver también Favilli
y Manganelli 2002).
La península ibérica no alberga ninguna especia nativa de
cangrejo de río (Ion et al. 2024). La hipótesis de una
introducción histórica del cangrejo italiano en España se había sugerido
durante décadas (por ejemplo, Albrecht 1983), pero
el evento de introducción original y el posterior proceso de invasión solo se
han descrito recientemente. El cangrejo italiano fue introducido para ser
criado en los jardines de los Sitios Reales, por deseo del rey Felipe II, quien
había negociado su adquisición durante al menos 25 años. Tras intentar obtener
cangrejos de río de Flandes (posiblemente Astacus astacus) y Francia (Austropotamobius
pallipes) en la década de 1560 (Clavero 2022),
el envío final se realizó desde la Toscana en 1588 (Clavero
et al. 2016). Este envío, bien documentado, explica de manera parsimoniosa
el hecho de que los cangrejos de río ibéricos pertenezcan a la subespecie Austropotamobius
fulcisianus subsp. fulcisianus (según Crandall y De Grave 2017), de la cual es sinónimo
Austropotamobius italicus subsp. italicus (Fratini et al. 2005; Pedraza-Lara
et al. 2010). La distribución nativa de esta subespecie se restringe a la
Toscana y regiones italianas vecinas (Jelić et al. 2016).
Más allá de la introducción documentada de Austropotamobius fulcisianus subsp. fulcisianus,
Clavero et al. (2016) revisaron evidencias generadas
por diferentes disciplinas, incluyendo patrones genéticos y filogeográficos,
para construir una narrativa sólida sobre el estatus no nativo del cangrejo
italiano en la península ibérica. Además de la evidencia genética, desarrollada
a continuación, el estudio destacó: i) las nociones implícitas y explícitas de
la ausencia de cangrejos de río en España antes de la introducción de 1588; ii)
la falta de un nombre vernáculo para el cangrejo de río en las lenguas
ibéricas; iii) el hecho de que el cangrejo de río se menciona por primera vez
como alimento en España en el contexto de la corte, a principios del siglo
XVII; iv) la ausencia de Branchiobdellida como ectosimbiontes (se habrían
perdido debido al transporte), una característica excepcional entre los
cangrejos de río europeos, compartida solo con los Austropotamobius pallipes
irlandeses, que habían sido introducidos desde Francia en época medieval (Gouin et al. 2003; Subchev 2014);
y v) el constante proceso de expansión de la especie debido a múltiples
introducciones secundarias, que continuó hasta los 1970s.
Los primeros estudios sobre la genética del género Austropotamobius
identificaron la conexión entre las poblaciones italianas e ibérica y
destacaron la baja diversidad genética esta últimas. Grandjean
et al. (2001) propusieron que estos patrones habrían surgido "por
un efecto fundador debido al origen antropogénico de los cangrejos de río en
España", aunque también consideraron otras posibilidades para explicar
la vicariancia ibérico-italiana. La principal alternativa era una hipotética
desaparición antigua (en el orden de decenas de miles de años) de A. fulcisianus
en el sur de Francia, debido a una supuesta expansión hacia el sur de A. pallipes
que, por una igualmente supuesta superioridad competitiva, habría desplazado a A.
fulcisianus, aislando poblaciones de las dos penínsulas. Este escenario no
tiene base biogeográfica, ya que todas las subespecies de A. fulcisianus
se encuentran confinadas, salvo introducciones, en la península itálica y la
costa adriática de la península balcánica, estando su distribución limitada por
las barreras que forman los Alpes y los Alpes Dináricos. Poco años después, Trontelj et al. (2005) concluyeron que "la
Península Ibérica ha sido repoblada artificialmente desde el norte de Italia",
siendo uno de los ejemplos "más evidentes" de la "influencia
masiva" que la propagación artificial ha tenido en la distribución de Austropotamobius
(ver también Gouin et al. 2003; Machino
et al. 2004).
Contradiciendo estas conclusiones, Diéguez-Uribeondo et al. (2008) afirmaron que
el "estatus nativo [de A. fulcisianus en la península
ibérica] no debería ser cuestionado", sentando las bases para
trabajos genéticos posteriores. Aun así, los autores no descartaron la
posibilidad de una introducción. Analizaron un fragmento del gen mitocondrial
COI y encontraron dos haplotipos en España, que diferían en una sola mutación,
uno de los cuales era exclusivo de España (es decir, no presente en poblaciones
italianas). Los propios autores reconocieron que el carácter privado del haplotipo
podría deberse a un fuerte sesgo de muestreo, ya que el estudio utilizó 229
individuos de 24 poblaciones españolas y solo 24 individuos de 4 poblaciones
italianas de A.
fulcisianus subsp. fulscianus. Lejos de ir
corrigiéndose, este sesgo de muestreo se ha acentuado notablemente en trabajos
posteriores (Fig. 2). Mientras que el tamaño de
la muestra ibérica ha aumentado constantemente, tanto en términos de
poblaciones como de individuos, no se ha hecho ningún esfuerzo por caracterizar
el acervo genético en Italia. Como resultado, la proporción de poblaciones italianas
incluidas en los diferentes estudios ha disminuido con el tiempo, cayendo por
debajo del 5 % en los artículos más recientes (Fig. 2).
Los trabajos genéticos que
presentan al cangrejo italiano como una especie nativa en la península ibérica
(Pedraza-Lara et al. 2010; Matallanas et al. 2011; 2016;
Martín-Torrijos et al. 2021; Martínez-Ríos et al. 2023) han basado sus
conclusiones en la riqueza y exclusividad de haplotipos detectados. Como se
discutió anteriormente, este razonamiento nos parece erróneo, y proponemos que
una correcta evaluación de las hipótesis en discusión debería centrarse en las
relaciones entre haplotipos. De hecho, Clavero et al.
(2016) y Clavero (2022) reinterpretaron los datos
publicados y demostraron que los patrones genéticos también tienden a confirmar
que la presencia de A. fulcisianus en la península ibérica se originó a
partir de una introducción desde Italia. En lo que sigue, utilizamos los
resultados del estudio más reciente (Martínez-Ríos
et al. 2023) para ejemplificar el respaldo genético a esta condición no
nativa, que contradice la interpretación de los autores originales. Dado que
los estudios genéticos tienden a progresar de manera anidada (es decir, los
estudios recientes utilizan las secuencias generadas por estudios previos,
añadiendo otras nuevas), centrarse en el trabajo más reciente permite una
discusión basada en un resumen de todas las contribuciones anteriores.
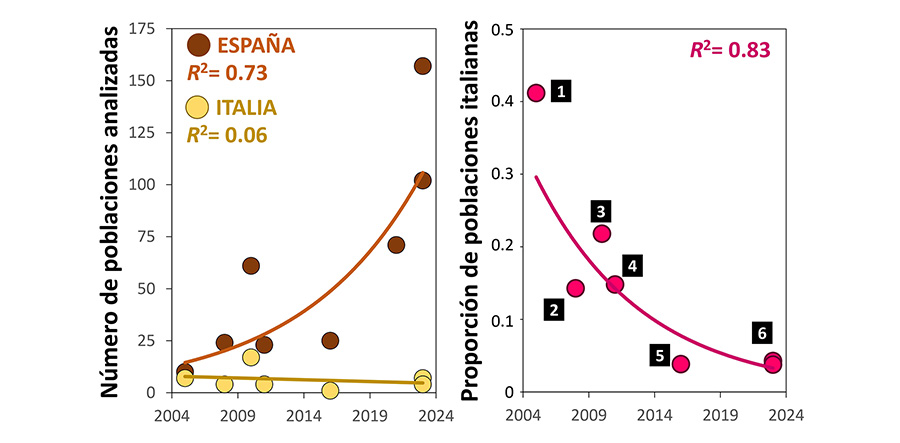
Figura 2. Poblaciones españolas e italianas de
cangrejo italiano (Austropotamobius fulcisianus) de la subespecie fulcisianus
utilizadas en estudios genéticos que tratan el estatus de la especie en la
península ibérica. El gráfico izquierdo muestra la evolución temporal del
número de poblaciones de ambos territorios, mientras el derecho muestra la
proporción de poblaciones italianas en los conjuntos de datos utilizados. Las
líneas y sus respectivos coeficientes de determinación (R²) corresponden
a funciones exponenciales. Los números en el panel izquierdo indican cada
estudio, como sigue: 1) Trontelj et al. (2005); 2) Diéguez-Uribeondo et al. (2008); 3) Pedraza-Lara et al. (2010); 4) Matallanas et al. (2011); 5) Matallanas et al. (2016); 6) Martínez-Ríos et al. (2023), con dos conjuntos de
datos (ver Figura 3).
Figure 2. Spanish and Italian Austropotamobius fulcisianus subsp. fulcisianus
populations used in genetic-based works dealing with the nativeness status of
the species in Iberia. Left panel shows the number of populations from both
territories in relation to the year of publication of the study, while right
one shows the proportion of Italian populations in datasets. Lines and their
respective coefficient of determination (R2) correspond to
exponential functions. Numbers in left panel indicate each study, as follows:
1) Trontelj et al. (2005); 2) Diéguez-Uribeondo et al. (2008);
3) Pedraza-Lara et
al. (2010); 4) Matallanas et al. (2011); 5) Matallanas et al.
(2016); 6) Martínez-Ríos et al. (2023), two datasets (see Figure 3).
Martínez-Ríos
et al. (2023) utilizaron dos conjuntos de datos, con secuencias
concatenadas de los genes 16S y COI, basados en información extraída de GenBank
y en sus propios trabajos de secuenciación. El primer conjunto de datos tenía
como objetivo maximizar la cobertura espacial y de linajes a expensas de
acortar la longitud de las secuencias (1344 individuos, 249 poblaciones,
secuencias de 948 pb para A. pallipes y A. fulcisianus), mientras
que el segundo priorizaba secuencias más largas de Austropotamobius
fulcisianus subsp. fulcisianus (661 individuos, 106 poblaciones,
secuencias de 2449 pb). Ambos conjuntos de datos tenían una representación
limitada de muestras italianas (ver punto 6 en Fig. 2),
a pesar de lo cual las redes de haplotipos resultantes siguen respaldando el
carácter introducido del cangrejo italiano en la península ibérica. Siguiendo
el razonamiento propuesto por Blakeslee et al. (2008), ejemplificado arriba con
los casos de Littorina o Hystrix, el hecho de que los haplotipos
detectados en España estén entremezclados en las redes con los detectados en
Italia (Fig. 3A y 3B)
es un indicador de que el cangrejo italiano fue introducido en España desde
Italia. Es verdad que ese patrón también sería compatible con que el cangrejo
italiano fuera nativo en la península ibérica y desde allí hubiese sido
introducido en Italia. Pero esta posibilidad no tiene, como se ha discutido
arriba, ninguna base biogeográfica y no se ajusta a los eventos históricos
conocidos. De hecho, la introducción desde Italia, y en concreto desde la
Toscana, concuerda con los resultados obtenidos a partir de otras
aproximaciones, destacando el hecho de que el conocimiento previo es
fundamental para contextualizar los resultados genéticos. Esto es evidente en
casos extremos, como los del ciervo corso o el linaje exclusivo de conejo en
Mallorca mencionados anteriormente, pero en todos los casos es un paso
necesario para construir narrativas filogeográficas o para desarrollar
hipótesis que puedan ser evaluadas explícitamente (ver Papadopoulou y Knowles 2016).
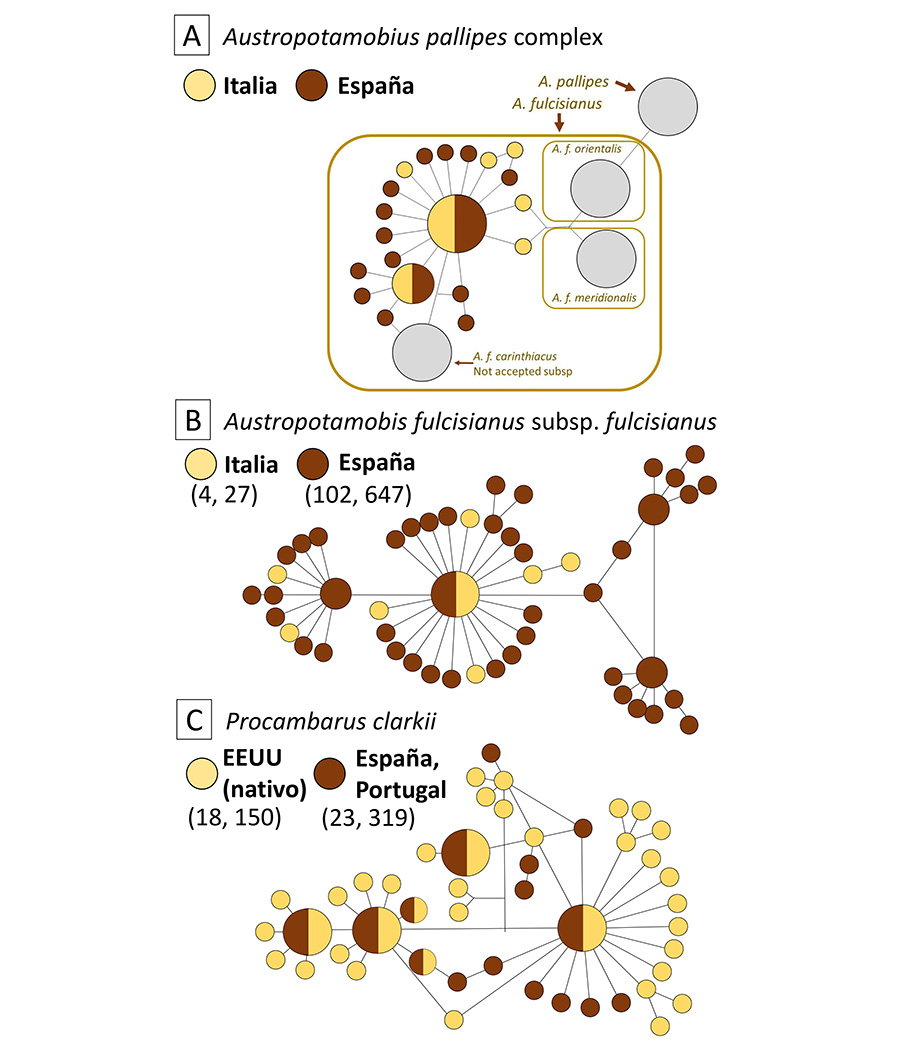
Figura 3. Relaciones entre los haplotipos de
ADN mitocondrial detectados en poblaciones ibéricas y nativas de Austropotamobius
(A, B, modificado de Martínez-Ríos et al.
2023) y Procambarus clarkii (C, modificado de Oficialdegui et al. 2019). A muestra una red de
haplotipos basada en una secuencia concatenada de 948 bp de los genes 16S y COI
para todo el complejo A. pallipes (que incluye a A. pallipes
propiamente dicho y A. fulcisianus), aunque solo se resaltan los
haplotipos correspondientes a A. fulcisianus subsp. fulcisianus
(la subespecie presente en Iberia). B presenta los resultados a partir de una
secuencia concatenada de 2449 bp de los mismos dos genes, cubriendo únicamente
muestras de A. fulcisianus subsp. fulcisianus. C representa las
relaciones entre un fragmento de 608 bp del gen COI de Procambarus clarkii.
Los valores entre paréntesis en B y C indican el número de poblaciones e
individuos analizados en cada una de las áreas (España/península ibérica vs.
área nativa).
Figure 3. Haplotype networks representing the relationships among
mitochondrial DNA haplotypes detected in Iberian and native populations of Austropotamobius
(A, B, modified from Martínez-Ríos et al.
2023) and Procambarus clarkii (C, modified from Oficialdegui et al. 2019). A shows a haplotype
network of a concatenated 948bp sequence of the 16S and COI genes for the
entire A. pallipes complex, although only haplotypes corresponding to A.
fulcisianus subsp. fulcisianus (the subspecies present in Iberia)
are highlighted. B results from a concatenated 2449 bp sequence of the same two
genes, covering only A. fulcisianus subsp. fulcisianus samples. C
represents the relationships among a 608 bp fragment of the COI gene of Procambarus
clarkii. Parentheses in B and C reflect the number of populations and
individuals analysed in each of the areas (Spain/Iberia vs native area).
Las redes y métricas de haplotipos observadas para Austropotamobius
en las penínsulas ibérica e itálica pueden compararse con los resultados de
análisis homólogos para el cangrejo rojo (Procambarus clarkii). Esta
especie, nativa del sureste de EE. UU. y el noreste de México, ha sido
introducida en más de 40 países en todo el mundo a lo largo de más de 100 años
(Oficialdegui et al. 2020). El cangrejo rojo
fue introducido en España en dos eventos, en 1973 y 1974, y se expandió
rápidamente por la península ibérica (Acevedo-Limón
et al. 2020). Oficialdegui et al. (2019)
analizaron un fragmento de 608 pb del gen COI en poblaciones de esta especie en
gran parte de su distribución actual. De las redes de haplotipos presentadas en
este trabajo, retuvimos únicamente los haplotipos presentes en poblaciones
nativas e ibéricas, resultando en redes notablemente similares a las del
cangrejo italiano (i.e., con una mezcla de haplotipos en áreas
nativas y no nativas) (Fig. 3). Al igual que en
el caso del cangrejo italiano, el cangrejo rojo en la península ibérica tiene
un gran número de haplotipos y alta variabilidad genética en algunas
poblaciones. Muchos de esos haplotipos (16) están compartidos con el área
nativa en EE. UU., pero hasta 10 de ellos se detectaron únicamente en la
península ibérica. Por supuesto, esta diversidad y exclusividad de haplotipos
no indica que el cangrejo rojo sea nativo en la península, como no lo hace en
el caso del cangrejo italiano. Patrones similares se conocen en otras especies
invasoras de cangrejos de río, como el cangrejo señal (Pacifastacus
leniusculus), una especie nativa del oeste de Norteamérica. Petrusek et al. (2017) reportaron una alta riqueza
haplotípica en poblaciones europeas de esta especie, con una mayoría de
haplotipos que no habían sido detectados en su área nativa. De nuevo, por
supuesto, esto no constituye ninguna evidencia de que el cangrejo señal sea nativa
en algún lugar de Europa.
Creemos que la supuesta alta diversidad haplotípica del
cangrejo italiano en la península ibérica, que Martín-Torrijos
et al. (2021) y Martínez-Ríos et al. (2023)
afirman haber desvelado —ambos artículos utilizan el término unraveling
en el título—, es en realidad un mero resultado del aumento en el esfuerzo de
muestreo, tanto en número de poblaciones (Fig. 2)
como en la longitud de los fragmentos de ADN analizados (comparar paneles A y B
en la Figura 3). Para equilibrar la comparación
entre las poblaciones italianas e ibéricas, el número de haplotipos debería
estandarizarse en función del tamaño de la muestra y la longitud de los
fragmentos de ADN. Aunque las mutaciones de ADN no ocurren con la misma frecuencia
en todas las regiones, calculamos el número de haplotipos estandarizado a 10
poblaciones y fragmentos de 1000 pb. Utilizando los fragmentos de ADN más
largos (Fig. 3B), estos valores son casi cinco
veces mayores en las poblaciones italianas (8.2) que en las españolas (1.7). De
hecho, la pérdida de diversidad haplotípica debido a las introducciones es
mayor en el cangrejo italiano que en el cangrejo rojo (32 y 9.3 en poblaciones
nativas e ibéricas, respectivamente).
En resumen, a pesar de la
alta riqueza y exclusividad haplotípica, sabemos que el cangrejo rojo no es
nativo en la península ibérica porque: 1) los patrones genéticos así lo
sugieren; 2) esto concuerda con el conocimiento biogeográfico; y,
especialmente, 3) conocemos las acciones y las motivaciones que llevaron a su
introducción en España, así como las fechas en las que ocurrió y las personas
que participaron en ella. Estas tres condiciones también se cumplen en el caso
del cangrejo italiano. Para ambas especies, la gran cantidad de conocimiento
acumulado respalda de manera sólida y coherente su estatus no nativo en la
península ibérica.
Conclusiones
En este trabajo mostramos cómo interpretaciones erróneas de
los patrones genéticos pueden generar, especialmente si se combinan con
muestreos sesgados, descripciones desencaminadas de eventos históricos y
patrones biogeográficos. Afortunadamente, existen pautas para interpretar los
resultados genéticos adecuadamente, centrando la atención en las relaciones
entre secuencias genéticas, lo que permite superar las limitaciones resultantes
de evaluar únicamente la riqueza y exclusividad de los haplotipos. Cuando estas
pautas se aplican al conflictivo caso del cangrejo italiano en España, queda
claro que el debate es artificial y que a día de hoy no existen evidencias que
cuestionen que Austropotamobius fulcisianus sea una especie introducida
(lo mismo que puede decirse de Procambarus clarkii). El cangrejo italiano
concentra una parte significativa de los esfuerzos dedicados a la conservación
de la biodiversidad de las aguas continentales en España (Alonso
2012), y recientemente se ha aprobado una estrategia nacional para su
conservación (MITECO 2024). Considerando la alta
proporción de especies endémicas y el pobre estado de conservación de la fauna
acuática ibérica (por ejemplo, Ribera 2000; Maceda-Veiga 2013; Nakamura
et al. 2023), resulta evidente que es necesario reevaluar las estrategias
de gestión y adaptarlas al hecho de que el cangrejo italiano sea una especie
introducida (Clavero 2014).
La conservación del cangrejo italiano en España se ha basado
en gran medida en la cría en cautividad y sueltas en zonas consideradas
propicias para establecer nuevas poblaciones, unas acciones a las que la
estrategia española pretende dar continuidad (MITECO 2024).
Los lugares de suelta tienen que ser sistemas de aguas permanentes y estar
libres de la presencia de especies americanas de cangrejos de río. Estos
hábitats son cada vez más escasos y son vitales para diversas especies, que
pueden verse perjudicados por la generación de nuevas poblaciones de cangrejos
de río (Rosewarne et al. 2013; James
et al. 2015). De hecho, tanto las especies americanas como las nativas
europeas de cangrejo de río tienen un enorme potencial de transformación de los
hábitats que ocupan (Gherardi et al. 2001; Weinländer y Füreder 2011; Nyström
2017), por lo que el impacto de las sueltas sobre la biodiversidad nativa
debería ser adecuadamente evaluado.
Desde una perspectiva más general, queremos resaltar el
valor del conocimiento interdisciplinar y los riesgos de valorar los resultados
de un enfoque particular por encima de otros. Cuando diferentes estudios
presentan resultados contradictorios, la generación de conocimiento sólido debe
basarse en una evaluación crítica de las fortalezas y debilidades de cada
metodología, sin asumir a priori la superioridad de ninguna en
particular. Al comunicar públicamente metodologías y resultados científicos,
tanto a la comunidad científica como a colectivos más amplios, es importante
reconocer que disciplinas con un componente tecnológico avanzado, como los
análisis genéticos, a menudo tienen mayor credibilidad que campos más
tradicionales. Como se ha demostrado en el caso del cangrejo italiano, tal
asunción puede no estar justificada. En el fomento de la cultura científica y
la divulgación del conocimiento es fundamental transmitir el valor de las
distintas disciplinas y las incertidumbres asociadas a cada una de ellas.
Financiación, permisos requeridos, potenciales
conflictos de interés y agradecimientos
Los autores/as declaran no tener ningún conflicto de
intereses.
Este trabajo es resultado del proyecto CRAYMAP (Crayfish invasions
across time and space, a multidisciplinary approach,
PID2020-20026RB-I00), financiado por el Ministerio de Ciencia e Innovación de
España. Queremos agradecer los comentarios de cuatro revisores anónimos y las
observaciones de los editores, que contribuyeron a mejorar este trabajo.
Contribución de los autores
MC, SB y FJO Conceptualización, Curación de datos,
Redacción- revisión y edición; MC Redacción- manuscrito original.
Disponibilidad de los datos
Este artículo no utiliza conjuntos de datos.
Referencias
Acevedo-Limón, L.,
Oficialdegui, F.J., Sánchez, M.I., Clavero, M. 2020. Historical,
human, and environmental drivers of genetic diversity in the red swamp crayfish
(Procambarus clarkii) invading the Iberian Peninsula. Freshwater
Biology 65: 1460-1474. https://doi.org/10.1111/fwb.13513
Albrecht, H. 1983. Besiedlungsgeschichte und ursprünglich holozane
verbreitung der europäischen Flusskrebse. Spixiana 6: 61-77.
Alcover, J.A. 2008. The first Mallorcans: prehistoric colonization in the
Western Mediterranean. Journal of World Prehistory 21, 19–84. https://doi.org/10.1111/fwb.13513
Alonso, F. 2011. Austropotamobius italicus (Faxon, 1914). Atlas
y libro rojo de los invertebrados amenazados de España (Especies Vulnerables)
(pp 651-672). Ministerio de Medio Ambiente Rural y Marino, Madrid.
Alonso, F. 2012. Austropotamobius
pallipes. Bases ecológicas preliminares para la conservación de las
especies de interés comunitario en España: Invertebrados (p. 69).
Ministerio de Agricultura, Alimentación y Medio Ambiente, Madrid.
Ascunce, M.S., Yang, C.C., Oakey,
J., Calcaterra, L., Wu, W.J., Shih, C-J., Goudet, J., et al. 2011. Global invasion history of the fire ant Solenopsis invicta. Science
331: 1066-1068. https://doi.org/10.1126/science.1198734
Baquero, R.A., Oficialdegui, F.J.,
Ayllón, D., Nicola, G.G. 2023. The challenge of managing
threatened invasive species at a continental scale. Conservation Biology
37: e14165. https://doi.org/10.1111/cobi.14165
Barbaresi, S., Gherardi, F., Mengoni, A., Souty-Grosset, C. 2007. Genetics
and invasion biology in fresh waters: a pilot study of Procambarus clarkii
in Europe. Biological invaders in inland waters: Profiles, distribution, and
threats (ed F. Gherardi) pp 381-400. Springer Dordrecht. The Netherlands. https://doi.org/10.1007/978-1-4020-6029-8_20
Bélouard, N., Paillisson, J.M., Oger, A., Besnard, A.L., Petit, E.J. 2019.
Genetic drift during the spread phase of a biological invasion. Molecular
Ecology 28: 4375-4387. https://doi.org/10.1111/mec.15238
Blackburn, T.M., Pyšek, P., Bacher, S., Carlton, J.T., Duncan, R.P.,
Jarošík, V., Wilson, J.R.U., et al. 2011. A proposed unified framework for
biological invasions. Trends in Ecology & Evolution 26: 333-339. https://doi.org/10.1016/j.tree.2011.03.023
Blackburn, T.M., Prowse, T.A., Lockwood, J.L., Cassey, P. 2013. Propagule
pressure as a driver of establishment success in deliberately introduced exotic
species: fact or artefact? Biological Invasions 15: 1459-1469. https://doi.org/10.1007/s10530-013-0451-x
Blakeslee, A.M. 2007. Native or invasive? The case history of the marine
snail, Littorina littorea, in northeast North America. In: Starkey, D.,
Holm, P., Barnard, M. (eds.), Oceans Past: Management Insights from the
History of Marine Animal Populations, pp. 7–24. Earthscan, London, UK.
Blakeslee, A.M., Byers, J.E., Lesser, M.P. 2008. Solving cryptogenic
histories using host and parasite molecular genetics: the resolution of Littorina
littorea's North American origin. Molecular Ecology 17: 3684-3696. https://doi.org/10.1111/j.1365-294X.2008.03865.x
Blakeslee, A.M., Altman, I., Miller, A.W., Byers, J.E., Hamer, C.E., Ruiz,
G.M. 2012. Parasites and invasions: a biogeographic examination of parasites
and hosts in native and introduced ranges. Journal of Biogeography 39:
609-622. https://doi.org/10.1111/j.1365-2699.2011.02631.x
Blumenfeld, A.J., Eyer, P.A., Husseneder, C., Mo, J., Johnson, L.N., Wang, C.,
Grace, J.K., et al. 2021. Bridgehead effect and multiple introductions shape
the global invasion history of a termite. Communications Biology 4: 196.
https://doi.org/10.1038/s42003-021-01725-x
Cabezas, M.P., Xavier, R., Branco, M., Santos, A.M., Guerra-García, J.M.
2014. Invasion history of Caprella scaura Templeton, 1836 (Amphipoda:
Caprellidae) in the Iberian Peninsula: multiple introductions revealed by
mitochondrial sequence data. Biological Invasions 16: 2221-2245. https://doi.org/10.1007/s10530-014-0660-y
Carlton, J.T. 1996. Biological invasions and cryptogenic species. Ecology
77: 1653-1655. https://doi.org/10.2307/2265767
Casas-Marce, M., Marmesat, E.,
Soriano, L., Martínez-Cruz, B., Lucena-Perez, M., Nocete, F.,
Rodríguez-Hidalgo, A., et al. 2017. Spatiotemporal dynamics of
genetic variation in the Iberian lynx along its path to extinction
reconstructed with ancient DNA. Molecular Biology and Evolution 34:
2893-2907. https://doi.org/10.1093/molbev/msx222
Clavero, M. 2014. Shifting baselines and the conservation of non-native
species. Conservation Biology 28: 1434-1436. https://doi.org/10.1111/cobi.12266
Clavero, M. 2016. Species substitutions driven by anthropogenic positive
feedbacks: Spanish crayfish species as a case study. Biological Conservation
193: 80-85. https://doi.org/10.1016/j.biocon.2015.11.017
Clavero, M. 2022. The King's aquatic desires: 16th century fish
and crayfish introductions into Spain. Fish and Fisheries 23: 1251-1263.
https://doi.org/10.1111/faf.12680
Clavero, M., Nores, C., Kubersky‐Piredda, S., Centeno‐Cuadros,
A. 2016. Interdisciplinarity to reconstruct historical introductions: solving
the status of cryptogenic crayfish. Biological Reviews 91:
1036-1049. https://doi.org/10.1111/brv.12205
Clavero, M., Suh, J., Franch, N.,
Aparicio, E., Buchaca, T., Caner, J., García-Rodriguez, S., et al. 2023. Invaders they are a-changing: A recent, unexpected surge of invasive
loaches in Catalonia. Freshwater Biology 68: 621-631. https://doi.org/10.1111/fwb.14051
Crain, P.K.,
Moyle, P.B. 2011. Biology, history, status and
conservation of Sacramento perch, Archoplites interruptus. San
Francisco Estuary and Watershed Science 9. https://doi.org/10.15447/sfews.2011v9iss1art5
Crandall, K.A., De Grave,
S. 2017. An updated classification of the freshwater
crayfishes (Decapoda: Astacidea) of the world, with a complete species list. The
Journal of Crustacean Biology 37: 615-653. https://doi.org/10.1093/jcbiol/rux070
Craven, D., Winter, M., Hotzel, K., Gaikwad, J., Eisenhauer, N., Hohmuth,
M., König-Ries, B., et al. 2019. Evolution of interdisciplinarity in
biodiversity science. Ecology and Evolution 9: 6744-6755. https://doi.org/10.1002/ece3.5244
Cunningham, C.W. 2008. How to use genetic data to distinguish between natural
and human-mediated introduction of Littorina littorea to North America. Biological
Invasions 10: 1-6. https://doi.org/10.1007/s10530-007-9099-8
Detry, C., Cardoso, J.L., Heras
Mora, J., Bustamante-Álvarez, M., Silva, A.M., Pimenta, J., Fernandes, I., et
al. 2018. Did the Romans introduce the Egyptian mongoose (Herpestes
ichneumon) into the Iberian Peninsula? The Science of Nature 105:
1-13. https://doi.org/10.1007/s00114-018-1586-5
Diéguez‐Uribeondo, J., Royo, F., Souty‐Grosset, C., Ropiquet, A.,
Grandjean, F. 2008. Low genetic variability of the white‐clawed
crayfish in the Iberian Peninsula: its origin and management implications. Aquatic
Conservation: Marine and Freshwater Ecosystems 18: 19-31. https://doi.org/10.1002/aqc.811
Díez-del-Molino, D.,
Sánchez-Barreiro, F., Barnes, I., Gilbert, M.T.P., Dalén, L. 2018. Quantifying temporal genomic erosion in endangered species. Trends
in Ecology & Evolution 33: 176-185. https://doi.org/10.1016/j.tree.2017.12.002
Dlugosch,
K.M., Parker, I.M. 2008. Founding events in species
invasions: genetic variation, adaptive evolution, and the role of multiple
introductions. Molecular Ecology 17: 431-449. https://doi.org/10.1111/j.1365-294X.2007.03538.x
Doan, K., Zachos, F.E., Wilkens, B., Vigne, J.D., Piotrowska, N.,
Stanković, A., Jędrzejewska, B., et al. 2017. Phylogeography of the Tyrrhenian
red deer (Cervus elaphus corsicanus) resolved using ancient DNA of
radiocarbon-dated subfossils. Scientific Reports 7: 2331. https://doi.org/10.1038/s41598-017-02359-y
Einfeldt, A.L., Doucet, J.R., Addison, J.A. 2014. Phylogeography and cryptic
introduction of the ragworm Hediste diversicolor (Annelida, Nereididae)
in the Northwest Atlantic. Invertebrate Biology 133: 232-241. https://doi.org/10.1111/ivb.12060
Einfeldt, A.L., Jesson, L.K., Addison, J.A. 2020. Historical human
activities reshape evolutionary trajectories across both native and introduced
ranges. Ecology and Evolution 10(13), 6579-6592. https://doi.org/10.1002/ece3.6391
Facon, B., Pointier, J.P., Jarne, P., Sarda, V., David, P. 2008. High
genetic variance in life-history strategies within invasive populations by way
of multiple introductions. Current Biology 18: 363-367. https://doi.org/10.1016/j.cub.2008.01.063
Favilli,
L., Manganelli, G. 2002. The Italian freshwater
crayfish (Austropotamobius fulcisianus) (Crustacea, Decapoda, Astacidae)
in the Farma-Merse basin (Southern Tuscany). Atti della Società Toscana di
Scienze Naturali Residente in Pisa. Memorie. Serie B 108: 43-49.
Faxon, W. 1914. Notes on the crayfishes in the United States National
Museum and the Museum of Comparative Zoology with descriptions of new species
and subspecies to which is appended a catalogue of the known species and
subspecies. Memoirs of the Museum of Comparative Zoölogy at Harvard College
40: 352–427. https://doi.org/10.5962/bhl.title.49092
Fischer, G.,
Fisher, B.L. 2013. A revision of Pheidole
Westwood (Hymenoptera: Formicidae) in the islands of the Southwest Indian Ocean
and designation of a neotype for the invasive Pheidole megacephala. Zootaxa
3683: 301-356. https://doi.org/10.11646/zootaxa.3683.4.1
Fok, K.W., Wade, C.M., Parkin, D.T. 2002. Inferring the phylogeny of
disjunct populations of the azure–winged magpie Cyanopica cyanus from
mitochondrial control region sequences. Proceedings of the Royal Society of
London. Series B: Biological Sciences 269: 1671-1679. https://doi.org/10.1098/rspb.2002.2057
Fratini, S., Zaccara, S., Barbaresi, S., Grandjean, F., Souty-Grosset, C.,
Crosa, G., Gherardi, F. 2005. Phylogeography of the threatened crayfish (genus Austropotamobius)
in Italy: implications for its taxonomy and conservation. Heredity 94:
108–118. https://doi.org/10.1038/sj.hdy.6800581
Gherardi, F., Renai, B., Corti, C. 2001. Crayfish predation on tadpoles: a
comparison between a native (Austropotamobius pallipes) and an alien
species (Procambarus clarkii). Bulletin Français de la Pêche
et de la Pisciculture 361: 659-668. https://doi.org/10.1051/kmae:2001011
Gippoliti,
S., Amori, G. 2006. Ancient introductions of
mammals in the Mediterranean Basin and their implications for conservation. Mammal
Review 36: 37-48. https://doi.org/10.1111/j.1365-2907.2006.00081.x
Gouin, N., Grandjean, F., Pain, S., Souty-Grosset, C. Reynolds, J. 2003.
Origin and colonization history of the white-clawed crayfish, Austropotamobius
pallipes, in Ireland. Heredity 91: 70–77. https://doi.org/10.1038/sj.hdy.6800282
Grandjean, F., Gouin, N., Souty-Grosset, C., Diéguez-Uribeondo, J. 2001.
Drastic bottlenecks in the endangered crayfish species Austropotamobius
pallipes in Spain and implications for its colonization history. Heredity
86: 431-438. https://doi.org/10.1046/j.1365-2540.2001.00849.x
Grandjean, F., Frelon-Raimond, M., Souty-Grosset, C. 2002. Compilation of
molecular data for the phylogeny of the genus Austropotamobius: one
species or several? Bulletin Francais de la Peche et de la
Pisciculture 367: 671-680. https://doi.org/10.1051/kmae:2002058
Hajji, G.M., Charfi-Cheikrouha, F.,
Lorenzini, R., Vigne, J.D., Hartl, G.B., Zachos, F.E. 2008. Phylogeography
and founder effect of the endangered Corsican red deer (Cervus elaphus
corsicanus). Biodiversity and Conservation 17: 659-673. https://doi.org/10.1007/s10531-007-9297-9
Haydar, D. 2012. What is natural? The scale of cryptogenesis in the North
Atlantic Ocean. Diversity and Distributions 18: 101-110. https://doi.org/10.1111/j.1472-4642.2011.00863.x
Hierro, J.L., Maron, J.L., Callaway, R.M. 2005. A biogeographical approach
to plant invasions: the importance of studying exotics in their introduced and
native range. Journal of Ecology 93: 5-15. https://doi.org/10.1111/j.0022-0477.2004.00953.x
Hofman, C.A.,
Rick, T.C. 2018. Ancient biological invasions and
island ecosystems: tracking translocations of wild plants and animals. Journal
of Archaeological Research 26: 65-115. https://doi.org/10.1007/s10814-017-9105-3
Hong, Y., Yuan, Z., Liu, X. 2024. Global drivers of the
conservation–invasion paradox. Conservation Biology e14290. https://doi.org/10.1111/cobi.14290
Hufbauer, R.A., Rutschmann, A., Serrate, B., Vermeil De Conchard, H., Facon,
B. 2013. Role of propagule pressure in colonization
success: disentangling the relative importance of demographic, genetic and
habitat effects. Journal of Evolutionary Biology 26: 1691-1699. https://doi.org/10.1111/jeb.12167
Hunter, M.E.,
Nico L.G. 2015. Genetic
analysis of invasive Asian Black Carp (Mylopharyngodon piceus) in the
Mississippi River Basin: evidence for multiple introductions. Biological
Invasions 17: 99-114. https://doi.org/10.1007/s10530-014-0708-z
Ion, M.C., Bloomer, C.C., Bărăscu, T.I., Oficialdegui, F.J., Shoobs,
N.F., Williams, B.W., Scheers, K., et al. 2024. World of Crayfish™: a web
platform towards real-time global mapping of freshwater crayfish and their
pathogens. PeerJ 12: e18229. https://doi.org/10.7717/peerj.18229
James, J., Slater, F.M., Vaughan, I.P., Young, K.A., Cable, J. 2015.
Comparing the ecological impacts of native and invasive crayfish: could native
species’ translocation do more harm than good? Oecologia 178:
309-316. https://doi.org/10.1007/s00442-014-3195-0
Jelić, M., Klobučar, G.I.,
Grandjean, F., Puillandre, N., Franjević, D., futo, M., Amouret, J., et al.
2016. Insights into the molecular phylogeny and historical
biogeography of the white-clawed crayfish (Decapoda, Astacidae). Molecular
Phylogenetics and Evolution 103, 26-40. https://doi.org/10.1016/j.ympev.2016.07.009
Kelager, A., Pedersen, J.S., Bruun, H.H. 2013. Multiple introductions and
no loss of genetic diversity: invasion history of Japanese Rose, Rosa rugosa,
in Europe. Biological Invasions 15: 1125-1141. https://doi.org/10.1007/s10530-012-0356-0
Klobučar, G.I., Podnar, M., Jelić, M., Franjević, D., Faller, M., Štambuk,
A., Gottstein, S., et al. 2013. Role of the Dinaric Karst (western Balkans) in
shaping the phylogeographic structure of the threatened crayfish Austropotamobius
torrentium. Freshwater Biology 58: 1089-1105. https://doi.org/10.1111/fwb.12110
Lambea-Camblor, A.,
Morcillo, F., Muñoz, J., Perdices, A. 2023. Genetic and
ecological approaches to introduced populations of pumpkinseed sunfish (Lepomis
gibbosus) in Southwestern Europe. Diversity 15: 1059. https://doi.org/10.3390/d15101059
Larroque, J., Chevret, P., Berger, J., Ruette, S., Adriaens, T., van den
Berge, K., Schockert, V., et al. 2023. Microsatellites and mitochondrial
evidence of multiple introductions of the invasive raccoon Procyon lotor
in France. Biological Invasions 25: 1955-1972. https://doi.org/10.1007/s10530-023-03018-2
Lejeusne, C., Saunier, A., Petit, N., Béguer, M., Otani, M., Carlton, J.T.,
Rico, C., et al. 2014. High genetic diversity and
absence of founder effects in a worldwide aquatic invader. Scientific
Reports 4: 5808. https://doi.org/10.1038/srep05808
Maceda-Veiga, A.
2013. Towards the conservation of freshwater fish:
Iberian Rivers as an example of threats and management practices. Reviews in
Fish Biology and Fisheries 23: 1-22. https://doi.org/10.1007/s11160-012-9275-5
Machino, Y., Füreder, L., Laurent, P.J., Petutschnig, J. 2004. Introduction
of the white-clawed crayfish Austropotamobius pallipes in Europe. Berichte
des naturwissenschaftlich-medizinischen Vereins in Innsbruck 91: 187-212.
Marchetti,
M.P., Engstrom, T. 2016. The conservation paradox
of endangered and invasive species. Conservation Biology 30: 434-437. https://doi.org/10.1111/cobi.12642
Martín-Torrijos, L., Buckley, D., Doadrio, I., Machordom, A., Diéguez-Uribeondo, J.
2021. Unraveling the hidden diversity of the native white claw crayfish in the
Iberian Peninsula. Frontiers in Ecology and Evolution 9: 669292. https://doi.org/10.3389/fevo.2021.669292
Martínez-Laiz, G., Ros, M., Guerra-García, J.M., Faasse, M., Santos, A.M.,
Cabezas, M.P. 2021. Using molecular data to monitor the post-establishment
evolution of the invasive skeleton shrimp Caprella scaura. Marine
Environmental Research 166: 105266. https://doi.org/10.1016/j.marenvres.2021.105266
Martínez-Ríos, M.,
Martín-Torrijos, L., Casabella-Herrero, G., Tedesco, P., Machordom, A.,
Diéguez-Uribeondo, J. 2023. On the conservation of
white-clawed crayfish in the Iberian Peninsula: Unraveling its genetic
diversity and structure, and origin. Plos One 18: e0292679. https://doi.org/10.1371/journal.pone.0292679
Masseti, M., Albarella, U.,
Mazzorin, J.D.G. 2010. The crested porcupine, Hystrix
cristata L., 1758, in Italy. Anthropozoologica 45: 27-42. https://doi.org/10.5252/az2010n2a2
Matallanas, B., Ochando, M.D.,
Vivero, A., Beroiz, B., Alonso, F., Callejas, C. 2011. Mitochondrial
DNA variability in Spanish populations of A. italicus inferred from the
analysis of a COI region. Knowledge and Management of Aquatic Ecosystems
401: 30. https://doi.org/10.1051/kmae/2011052
Matallanas, B., Ochando, M.D.,
Alonso, F., Callejas, C. 2016. Update of genetic information
for the white-clawed crayfish in Spain, with new insights into its population
genetics and origin. Organisms Diversity & Evolution 16:
533-547. https://doi.org/10.1007/s13127-016-0268-4
MITECO 2024. Estrategia para la
conservación del cangrejo de río ibérico (Austropotamobius pallipes) en
España. Disponible en: https://www.miteco.gob.es/content/dam/miteco/es/biodiversidad/publicaciones/estrategias/Estrategia-cangrejo-25-1.pdf.
Muirhead, J.R., Gray, D.K.,
Kelly, D.W., Ellis, S.M., Heath, D.D., Macisaac, H.J. 2008. Identifying
the source of species invasions: sampling intensity vs. genetic diversity. Molecular
Ecology 17: 1020-1035. https://doi.org/10.1111/j.1365-294X.2008.03669.x
Nakamura, K., Sousa, R.,
Mesquita-Joanes, F. 2023. Collapse of native freshwater mussel
populations: Prospects of a long-term study. Biological Conservation
279: 109931. https://doi.org/10.1016/j.biocon.2023.109931
Ninni, A.P. 1886. Sul gambero
fluviale italiano. Atti della Società Italiana di Scienze Naturali 29:
323–326.
Nyström, P. 2017. Ecological impact of introduced and native crayfish on
freshwater communities: European perspectives. In: Gherardi, F., Holdich, D.M.
(eds.), Crayfish in Europe as alien species, pp. 63-85. Routledge. https://doi.org/10.1201/9781315140469-6
Oficialdegui, F.J., Clavero, M., Sánchez, M.I., Green, A.J., Boyero, L., Michot,
T.C., Klose, K., et al. 2019. Unravelling the global invasion routes of a
worldwide invader, the red swamp crayfish (Procambarus clarkii). Freshwater
Biology 64: 1382-1400. https://doi.org/10.1111/fwb.13312
Oficialdegui, F.J., Sánchez, M.I., Clavero, M. 2020. One century away from home:
how the red swamp crayfish took over the world. Reviews in Fish Biology and
Fisheries 30: 121-135. https://doi.org/10.1007/s11160-020-09594-z
Oficialdegui, F.J., South, J., Courchamp, F., Clavero, M. 2024. Nativeness is a
binary concept—Invasiveness and its management are not. Biological
Conservation 294: 110631. https://doi.org/10.1016/j.biocon.2024.110631
Papadopoulou,
A., Knowles, L.L. 2016. Toward a paradigm shift in
comparative phylogeography driven by trait-based hypotheses. Proceedings of
the National Academy of Sciences 113: 8018-8024. https://doi.org/10.1073/pnas.1601069113
Pârvulescu, L. 2019. Introducing a new Austropotamobius crayfish
species (Crustacea, Decapoda, Astacidae): A miocene endemism of the Apuseni
Mountains, Romania. Zoologischer Anzeiger 279: 94-102. https://doi.org/10.1016/j.jcz.2019.01.006
Pedraza-Lara, C., Alda, F.,
Carranza, S., Doadrio, I. 2010. Mitochondrial DNA structure of
the Iberian populations of the white-clawed crayfish, Austropotamobius
italicus italicus (Faxon, 1914). Molecular Phylogenetics and Evolution
57: 327-342. https://doi.org/10.1016/j.ympev.2010.06.007
Peters, J., Lebrasseur, O., Irving-Pease, E.K., Paxinos, P.D., Best, J.,
Smallman, R., Callou, C., et al. 2022. The biocultural origins and dispersal of
domestic chickens. Proceedings of the National Academy of Sciences 119:
e2121978119. https://doi.org/10.1073/pnas.2121978119
Petrusek, A., Filipová, L.,
Kozubíková-Balcarová, E., Grandjean, F. 2017. High genetic
variation of invasive signal crayfish in Europe reflects multiple introductions
and secondary translocations. Freshwater Science 36: 838-850. https://doi.org/10.1086/694866
Pipek, P., Pysek, P., Blackburn, T.M. 2015. How the Yellowhammer became a
Kiwi: the history of an alien bird invasion revealed. Neobiota 24: 1-31.
https://doi.org/10.3897/neobiota.24.8611
Pooley, S.P., Mendelsohn, J.A., Milner-Gulland, E.J. 2014. Hunting down
the chimera of multiple disciplinarity in conservation science. Conservation Biology 28: 22-32. https://doi.org/10.1111/cobi.12183
Rosewarne, P.J., Mortimer, R.J.G., Dunn, A.M. 2013. Size-dependent impacts of
the endangered white-clawed crayfish (Austropotamobius pallipes) (Lereboullet)
on the littoral community. Knowledge and Management of Aquatic Ecosystems
409: 06. https://doi.org/10.1051/kmae/2013047
Ribera, I. 2000. Biogeography and conservation of Iberian water beetles. Biological
Conservation 92: 131-150. https://doi.org/10.1016/S0006-3207(99)00048-8
Rius, M.,
Darling, J.A. 2014. How important is intraspecific
genetic admixture to the success of colonising populations? Trends in
Ecology & Evolution 29: 233–242. https://doi.org/10.1016/j.tree.2014.02.003
Robin, M., Ferrari, G., Akgül, G., Münger, X., von Seth, J., Schuenemann,
V.J., Dalén, L., et al. 2022. Ancient mitochondrial and modern whole genomes
unravel massive genetic diversity loss during near extinction of Alpine ibex. Molecular
Ecology 31: 3548-3565. https://doi.org/10.1111/mec.16503
Roman, J.,
Darling, J.A. 2007. Paradox lost: genetic diversity
and the success of aquatic invasions. Trends in Ecology & Evolution
22: 454-464. https://doi.org/10.1016/j.tree.2007.07.002
Seebens, H., Blackburn, T.M., Dyer, E.E., Genovesi, P., Hulme, P.E.,
Jeschke, J.M., Pagad, S., et al. 2017. No saturation in the accumulation of
alien species worldwide. Nature Communications 8: 14435. https://doi.org/10.1038/ncomms14435
Seixas, F.A., Juste, J., Campos,
P.F., Carneiro, M., Ferrand, N., Alves, P.C., Melo-Ferreira, J.M. 2014. Colonization history of Mallorca Island by the European rabbit, Oryctolagus
cuniculus, and the Iberian hare, Lepus granatensis (Lagomorpha:
Leporidae). Biological Journal of the Linnean Society 111: 748-760. https://doi.org/10.1111/bij.12248
Sequeira, F., Aguilar, F.F.,
Madeira, F.M., Teixeira, J., Crespo, E., Ferrand, N., Rebelo, R. 2022. What can genetics tell us about the history of a human-mediated
introduction of the golden-striped salamander south of its native range? European
Journal of Wildlife Research 68: 78. https://doi.org/10.1007/s10344-022-01628-0
Subchev, M. 2014. The genus Branchiobdella Odier, 1823 (Annelida,
Clitellata, Branchiobdellida): a review of its European species. Acta
Zoologica Bulgarica 66: 5–20.
Szabó, P. 2010. Why history matters in ecology: an interdisciplinary
perspective. Environmental Conservation 37: 380-387. https://doi.org/10.1017/S0376892910000718
Tedeschi, L., Lenzner, B., Schertler, A., Biancolini, D., Essl, F.,
Rondinini, C. 2024. Threatened mammals with alien populations: distribution,
causes, and conservation. Conservation Letters: e13069. https://doi.org/10.1111/conl.13069
Trontelj, P., Machino, Y., Sket, B. 2005. Phylogenetic and phylogeographic
relationships in the crayfish genus Austropotamobius inferred from
mitochondrial COI gene sequences. Molecular Phylogenetics and Evolution
34: 212-226. https://doi.org/10.1016/j.ympev.2004.09.010
Trucchi, E.,
Sbordoni, V. 2009. Unveiling an ancient biological
invasion: molecular analysis of an old European alien, the crested porcupine (Hystrix
cristata). BMC Evolutionary Biology 9: 1-10. https://doi.org/10.1186/1471-2148-9-109
Uller, T., Leimu, R. 2011. Founder events predict changes in genetic diversity
during human-mediated range expansions. Global Change Biology 17:
3478-3485. https://doi.org/10.1111/j.1365-2486.2011.02509.x
van Boheemen, L.A., Lombaert, E., Nurkowski, K.A., Gauffre, B., Rieseberg, L.H.,
Hodgins, K.A. 2017. Multiple introductions, admixture and bridgehead invasion
characterize the introduction history of Ambrosia artemisiifolia in
Europe and Australia. Molecular Ecology 26: 5421-5434. https://doi.org/10.1111/mec.14293
Weinländer, M., Füreder,
L. 2011. Crayfish as trophic agents: effect of Austropotamobius
torrentium on zoobenthos structure and function in small forest streams. Knowledge
and Management of Aquatic Ecosystems 401: 22. https://doi.org/10.1051/kmae/2011039
Wilmshurst, J.M., Anderson, A.J., Higham, T.F., Worthy, T.H. 2008. Dating the
late prehistoric dispersal of Polynesians to New Zealand using the commensal
Pacific rat. Proceedings of the National Academy of Sciences 105:
7676-7680. https://doi.org/10.1073/pnas.0801507105
![]() , Sergio Bedmar1
, Sergio Bedmar1 ![]() , Francisco J. Oficialdegui2
, Francisco J. Oficialdegui2 ![]()


